日本マウスクリニック支援技術
Genome scanning
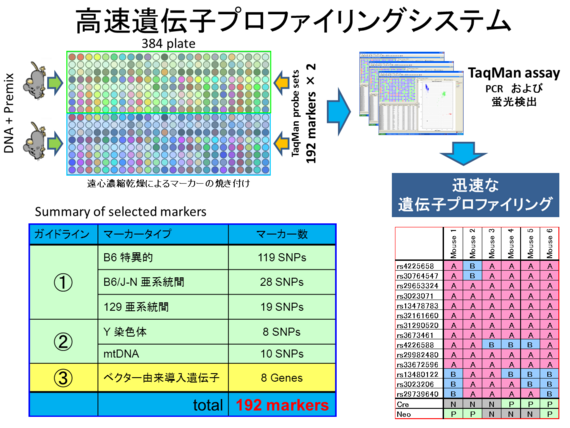
解析対象のマウス系統において、不均一な遺伝背景は表現型に影響を与える可能性があります。そのため、均一な遺伝背景を持った個体群を用いて表現型解析を行うことが望ましいと考えられます。そこで我々は、遺伝背景を検査することが可能な高速遺伝子プロファイリングシステムを構築し、クライアントから送付されてきた個体に対するWhole genome scanningを実施しています。システムは384プレートベースのTaqManアッセイ法を用いることで、迅速性と簡便性を両立させています。また、詳細な遺伝背景を検出するために、システムの中で使用する192のマーカーは、以下の3つのガイドラインに基づいて選択されています。
ガイドライン①:
ゲノムワイドに遺伝背景の均一性やコンジェニック化の程度などを検出するために、常染色体やX染色体上に存在する計166のSNPsマーカーが選択されています。これらの内訳は、C57BL/6系統への戻し交配を検出するためにC57BL/6系統がその他の多くのマウス系統に対して多型をもつ119 SNPsとC57BL/6の亜系統を見極めるためのC57BL/6JとC57BL/6Nの間の28 SNPs (C57BL/6Jブリーダー間のSNPsを若干含む) (目加田ら 2009)と各種ES細胞の系統として用いられる129亜系統間からの19 SNPsから構成されています。
ガイドライン②:
父系・母系を検出する目的で計18のSNPsマーカーがY染色体とミトコンドリアDNA上から選択されています。どちらかの性別のキャリアーを一方的に選択して戻し交配を行った結果、しばしば、これらは未置換の場合があるためです。
ガイドライン③:
遺伝子改変系統作製の際に利用されるneo、cre、Gfp等のベクター由来導入遺伝子の検出するためのマーカーに相当しています(中田ら 2009)。導入遺伝子の慮外の残存や不完全なKOアレルの構築などを検査するためです。
システムの基本デザイン
384プレートの各ウエルに2セットの192マーカーをあらかじめ濃縮遠心乾燥しておきます。1個体分のDNAとTaqMan反応液からなる混合液の2個体分を各ウエルに分注し、PCRと蛍光検出するだけでよいTaqManアッセイ法の特徴を生かすことによって、迅速かつ簡便に個体レベルでの遺伝子プロファイリングを出力することが可能になっています。
参考文献
1. Genetic differences among C57BL/6 substrains. Mekada K, Abe K, Murakami A,
Nakamura S, Nakata H, Moriwaki K, Obata Y, Yoshiki A. Exp Anim.
2009 Apr;58(2):141-9.
2. Simultaneous detection of multiple transgenes for genetically-modified mouse strains.
Nakata H, Hashimoto T, Seki Y, Mekada K, Obata Y, Yoshiki A. Exp Anim.
2009 Jul;58(4):437-42.

